Antibiogramme des mycobactéries tuberculeuses
Les mycobactéries du complexe tuberculosis sont naturellement résistantes aux antibiotiques utilisés classiquement à l’exception des aminosides, des rifamycines, et des fluoroquinolones. Parmi les antibiotiques susceptibles d’être actifs (les antituberculeux) citons : l’isoniazide( INH), l’éthambutol (EMB), la rifampicine (RMP) et la streptomycine (SM), la pyrazinamide (PYZ).
Remarque : M. bovis est naturellement résistant au pyrazinamide.
La résistance acquise aux antibiotiques chez les mycobactéries tuberculeuses est toujours liée à des mutations chromosomiques.
Dans une population bactérienne d’une souche dite sensible à un antibiotique, il existe toujours un faible pourcentage (<1%) de bacilles mutants et donc résistants.
Si ce pourcentage dépasse 1%, la souche est alors catégorisée « résistante ».
Pour éviter la sélection de ces individus résistants, il est nécessaire d’associer les antibiotiques antituberculeux.
L’antibiogramme des mycobactéries peut se faire :
- soit par méthode phénotypique en étudiant le comportement des mycobactéries en présence de divers antibiotiques, on distingue alors les méthodes en milieu solide (méthodes des proportions) et les méthodes en milieu liquide.
- soit par méthodes génotypiques en utilisant des sondes capables de détecter les mutations à l’origine des résistances.
Antibiogramme par la méthode des proportions
Cette méthode permet de mesurer la proportion de bacilles capables de donner des colonies en présence d’un antibiotique à une concentration donnée.
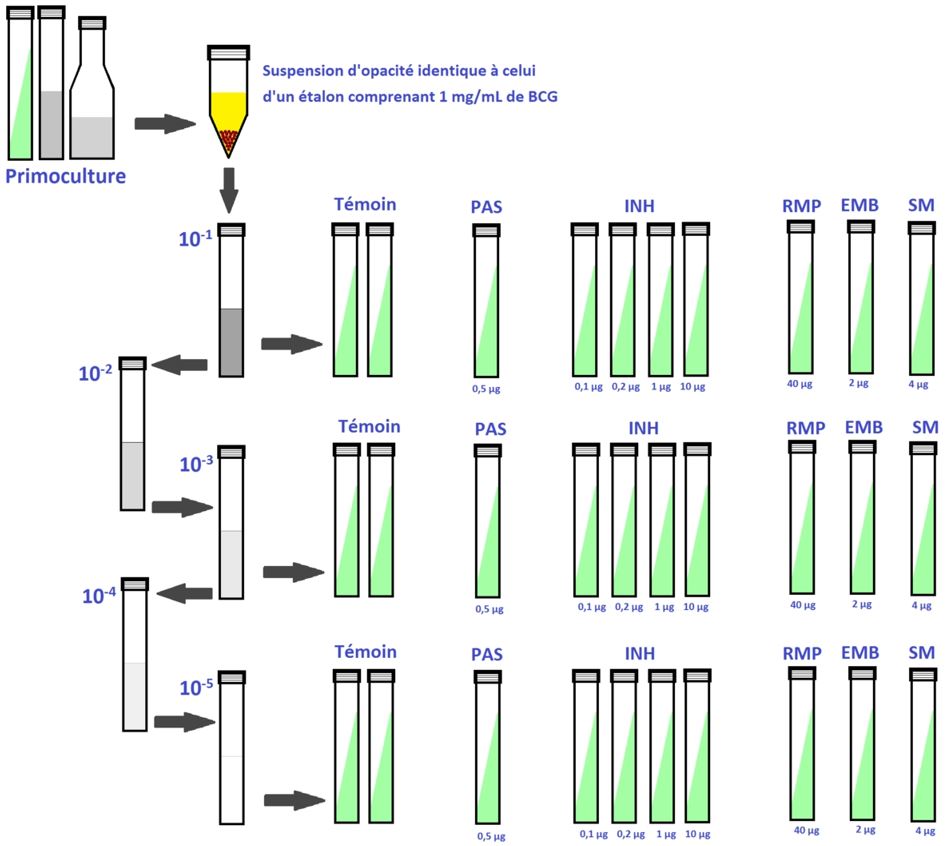
Ensemencement à partir d’une culture sur milieu solide
Trois dilutions de la souche étudiée sont ensemencées sur une série de tubes comprenant :
- deux milieux Löewenstein Jensen témoins (= sans antibiotique) ;
- d’autres milieux Löewenstein Jensen comprenant un antibiotique à une concentration donnée.
La suspension de départ est obtenue en déposant de 5 à 10 colonies dans un tube contenant des billes de verre et 0,5 mL d’eau distillée stérile. Après homogénéisation, cette suspension est ensuite ajustée en ajoutant de l’eau distillée stérile de façon à présenter un trouble d’opacité identique à celui d’un étalon comprenant 1 mg/mL de BCG.
Trois dilutions (10-1, 10-3, 10-5) sont réalisées et les séries de tubes sont ensemencées avec 0,2 mL. Les milieux sont ensuite incubés à 37°C.
On effectue une première lecture après 3 semaines et une lecture définitive après 6 semaines.
Exemple de résultats d’antibiogramme en milieu solide d’une souche de BK (son interprétation dans le cadre grisé ci-dessous)
|
Dilution |
Nombre de colonies |
|||
|
Témoin |
Rifampicine |
Ethambutol |
Streptomycine |
|
|
10-1 |
incomptable |
3 colonies |
Pas de colonie |
incomptable |
|
10-3 |
20 colonies |
Pas de colonie |
Pas de colonie |
1 colonie |
|
10-5 |
Pas de colonie |
Pas de colonie |
Pas de colonie |
Pas de colonie |
Ensemencement à partir d’une culture en bouillon MGIT
Il est également possible de pratiquer un antibiogramme en milieu solide par la méthode des proportions à partir d’une culture en bouillon MGIT. Cependant pour que le nombre de mycobactéries soit suffisant, il faut disposer d’une culture en MGIT détectée positive depuis 3 à 5 jours.
Dans ce cas, seule deux séries de tubes sont ensemencées, une avec une dilution au 1/5 et une dilution au 1/50 en eau physiologique de la culture en bouillon MGIT. Comme précédemment on ensemence chaque tube avec 0,2 mL.
Antibiogramme en milieu liquide
Exemple : utilisation de l’automate BACTEC MGIT 960
Il est pratiqué à partir de culture en bouillon MGIT.
Des milieux MGIT contenant chacun un antibiotique et un milieu MGIT témoin sans antibiotique sont ensemencés. L’inoculum du témoin est une dilution au 1/100 de l’inoculum des milieux avec antibiotique (sauf pour le pyrazinamide : 1/10).
Une souche est considérée sensible à un antibiotique donné si dans les 48 heures qui suivent la positivité du témoin, aucune fluorescence n’est détectée dans le milieu contenant cet antibiotique.
En effet, le témoin contenant 100 fois moins de bactérie que les milieux avec antibiotique, si le pourcentage de bactéries résistantes est égal à 1%, la culture dans les tubes avec antibiotique devrait être positive en même temps que le témoin.
Ainsi, lorsque dans les 48 heures qui suivent la positivité du témoin, aucune fluorescence n’est détectée dans le milieu contenant cet antibiotique, on peut conclure que le % de bactéries résistantes est inférieur à 1% (10% pour le pyrazinamide), la souche sera considérée alors sensible.
Cette méthode est plus rapide, il faut entre 5 à 12 jours pour obtenir l’antibiogramme d’une mycobactérie tuberculeuse.
Méthodes génotypiques de détection de la résistance
Elles reposent sur la détection des mutations affectant les gènes codant pour la cible des antibiotiques.
La résistance à la rifampicine résulte d’une mutation dans le gène rpoB qui code pour l’ARN polymérase ADN dépendante.
La résistance à l’isoniazide fait suite à des mutations touchant divers gènes (katG qui code pour une catalase, inhA qui code pour une enzyme impliquée dans la synthèse des acides mycoliques, ahpC qui code pour une protéine impliquée dansla réponse au stress oxydatif).
Les régions de ces gènes le plus fréquemment touchées par ces mutations sont amplifiées puis les amplicons sont détectés par hybridation voire séquençage.
Parmi les trousses commercialisées, citons :
- INNOLiPA Rif Tb pour la résistance à la rifampicine (sensibilité de 90 à 97% et spécificité proche de 100%) ;
- GenoType® MTBDR : sensibilité de 99% pour la rifampicine et 88,4 % pour l ’INH.
GenoType® MTBDR
Fiche technique du GenoType® MTBDR
Avec ce test, on utilise deux procédés pour la mise en évidence des mutations.
- Reconnaissance de la nouvelle séquence par des sondes « mutation » comme rpoB mutation probe 1
- Non reconnaissance d’une séquence présente sur les souches sauvages par des sondes « sauvages » comme rpoB wild type probe 1
Comme il est impossible de disposer de toutes les sondes permettant de reconnaître les mutations, on utilise des sondes capables de reconnaître une séquence de la souche sauvage. Quand l’amplicon est incapable de s’hybrider à cette sonde, cela signifie qu’une mutation est apparue sur la région qui aurait du s’hybrider à la sonde.
Les séquences correspondant à ces mutations sont amplifiées en utilisant des sondes biotinylées puis les amplicons sont détectés par hybridation inverse.
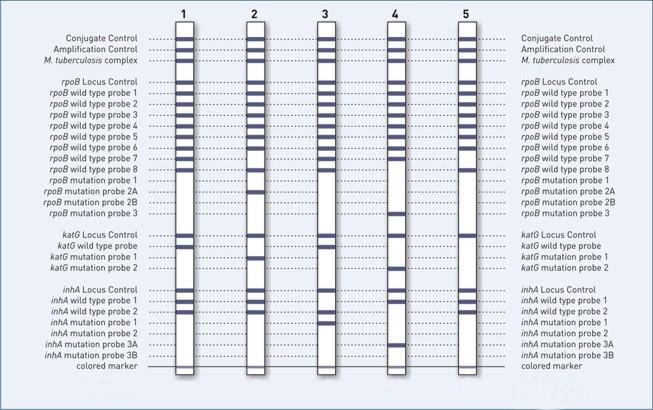
Pour faciliter la compréhension de l’interprétation de ce test, cinq résultats obtenus avec des souches différentes de mycobactéries tuberculeuses sont représentés ci dessous.
INTERPRÉTATION
La souche 1 est sensible. Pas d’hybridation des sondes « mutations » et hybridation de toutes les sondes « wild » (sauvage).
La souche 2 présente diverses mutations qui lui confèrent une résistance à la rifampicine et l’isoniazide.
- Résistance à la rifampicine car :
- mise en évidence d’une mutation par la sonde rpoB mutation probe 2a = apparition d’une bande
- mise en évidence d’une mutation par la sonde rpoB wild type probe 7 = disparition d’une bande
- Résistance à l’isoniazide car :
- mise en évidence d’une mutation par la sonde katG mutation probe 1 = apparition d’une bande
- mise en évidence d’une mutation par la sonde katG wild type probe = disparition d’une bande
La souche 3 résiste à l’isoniazide car :
- mise en évidence d’une mutation par la sonde inhA mutation probe 1 = apparition d’une bande
- mise en évidence d’une mutation par la sonde inhA wild type probe 1 = disparition d’une bande
La souche 4 présente diverses mutations qui lui confèrent une résistance à la rifampicine et l’isoniazide.
- Résistance à la rifampicine car :
- mise en évidence d’une mutation par la sonde rpoB mutation probe 3 = apparition d’une bande
- mise en évidence d’une mutation par la sonde rpoB wild type probe 8 = disparition d’une bande
- Résistance à l’isoniazide car :
- mise en évidence d’une mutation par la sonde katG mutation probe 2 = apparition d’une bande
- mise en évidence d’une mutation par la sonde katG wild type probe = disparition d’une bande
- mise en évidence d’une mutation par la sonde inhA mutation probe 3A = apparition d’une bande
- mise en évidence d’une mutation par la sonde inhA wild type probe 2 = disparition d’une bande
La souche 5 présente diverses mutations qui lui confèrent une résistance à la rifampicine et l’isoniazide.
- Résistance à la rifampicine car :
- mise en évidence d’une mutation par la sonde rpoB wild type probe 7 = disparition d’une bande
- Résistance à l’isoniazide car :
- mise en évidence d’une mutation par la sonde katG wild type probe = disparition d’une bande