Coproculture
Un enfant de 3 ans souffre de diarrhées et présente une fièvre modérée et des douleurs abdominales. Il n’a pas voyagé récemment.
Son médecin prescrit une coproculture standard.
| Aspect des selles |
Selles pâteuses malodorantes avec des traces de sang |
| Gram objectif X100 |
Flore comprenant 90% de petits bacilles à Gram négatif et 10% de bactéries à Gram positif |
| EF objectif x40 |
Leucocytes : environ 12 /champ
Hématies : 6 /champs Pas de mobilité en vol de moucheron, pas de levures |
| BCP à 37°C | Nombreuses petites colonies lactose -. Quelques colonies lactose + |
| Hektoen à 37°C | Présence de deux types de colonies. De nombreuses petites colonies de 0,5 mm de diamètre et quelques colonies de 2 à 3 mm de diamètre. Ces deux types sont de couleur saumon. |
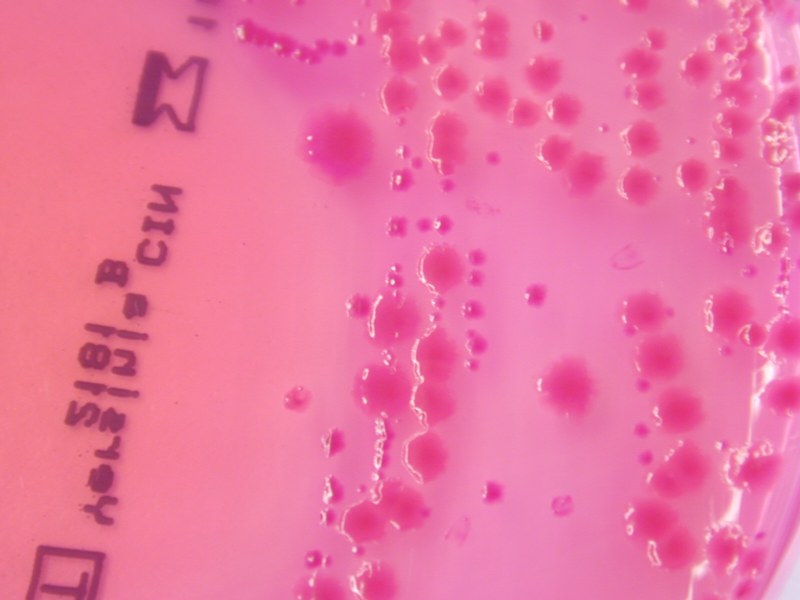
| Yersinia CIN à 30°C | Présence de petites colonies rouges et de grosses colonies rouges.
|
| CAMPYLOSEL à 37°C sous atmosphère microaérobie | Pas de culture |
| Bouillon Mueller Kauffman | Présence d’un trouble |
Questions
| Pour faire apparaitre la réponse, cliquez sur la question ! |
La flore est déséquilibrée.
La présence de leucocytes oriente vers une diarrhée inflammatoire.
Suspicion d’une infection invasive par le bacille à Gram négatif dominant.
Il ne semble pas qu’il y ait des Campylobacter ou des Vibrio car il n’a pas été observé de bactérie présentant une mobilité « en vol de moucheron ». Mais cela reste à confirmer en poursuivant la recherche des Campylobacter par la culture. En revanche, l’ensemencement de milieux sélectifs des Vibrio n’a pas été jugé nécessaire car il n’y a pas de notion de voyage récent.
| Liens utiles |
| Coproculture |
Le bacille Gram négatif dominant semble former les petites colonies lactose – sur BCP.
Les colonies saumon sur Hektoen utilisent au moins un des 3 glucides, ce ne sont pas des Salmonella, ni des Shigella.
Les deux types de colonies sur Yersinia CIN sont rouges donc mannitol +. Ces colonies sont suspectes d’être des Yersinia.
Isolement sur milieu chromogène sélectif des Salmonella, la culture en bouillon Rappaport-Vassiliadis.
Éventuellement un tests uréase rapide avec les deux types de colonies rouges sur Yersinia CIN.
Poursuite de l’identification des 2 types de colonies rouges (avec API 20E par exemple)
Remise à l’étuve du milieu Campylosel.
| Les tests uréase réalisés sur les deux types de colonies sont négatifs. |
| Pas de culture sur Campylosel et présence seulement de colonies bleues sur le milieu Rambach. |
Effectivement les Yersinia enterocolitica et pseudotuberculosis sont uréase +. Cependant il ne serait pas prudent d’écarter les Yersinia avec un test négatif, d’autant plus quand il s’agit d’un test rapide. L’intérêt de ce test est relatif puisque l’analyse est poursuivie quel que soit son résultat.
|
Lecture du contrôle de pureté = un seul type de colonie |
. . On observe un seule type de colonie sur le contrôle de pureté, la suspension utilisée pour ensemencer la galerie n’a pas été contaminée.
| Document utile |
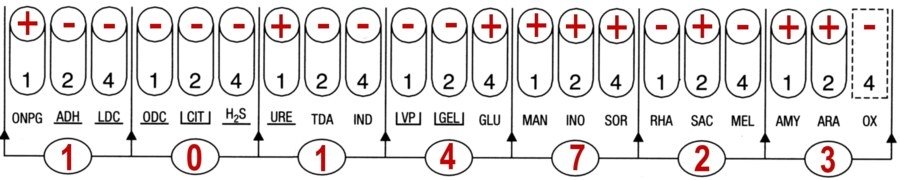
 Fiche technique de la galerie API 20E |
La galerie API 20E présente un profil caractéristique de Yersinia enterocolitica.
| Liens utiles |
| Identifieur « api » d’UPBM le Lab (un grand merci à Antoine Gaudin et Jean-Noël Joffin !) |
|
Lecture du contrôle de pureté = un seul type de colonie |
On observe un seule type de colonie sur le contrôle de pureté, la suspension utilisée pour ensemencer la galerie n’a pas été contaminée.
| Document utile |
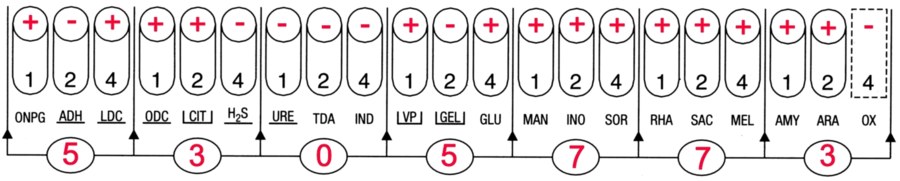
 Fiche technique de la galerie API 20E |
La galerie API 20E présente un profil caractéristique de Enterobacter aerogenes.
| Liens utiles |
| Identifieur « api » d’UPBM le Lab (un grand merci à Antoine Gaudin et Jean-Noël Joffin !) |
Ensemencer les milieux suivants avec la souche de Yersinia enterocolitica :
gélose à l’esculine
un Hugh et Leifson + Xylose à 1%
un Hugh et Leifson + Tréhalose à 1%.
Cette galerie permettra de connaitre le biotype de la souche.
| La souche apparaît esculine – ; xylose – et tréhalose + |
La souche est une Yersinia enterocolitica de biotype 4. C’est un biotype pathogène.
Diarrhée due à Yersinia enterocolitica biotype 4
| Liens utiles |
| Identification des biotypes de Yersinia enterocolitica |
Ce patient présente une diarrhée causée par une souche de Yersinia enterecolitica de biotype 4.
La souche est envoyée à l’Institut Pasteur pour vérifier le biotype et déterminer son sérotype. Le médecin doit déclarer ce cas de TIAC aux autorités sanitaires.
Les colonies bleues sur Rambach sont Propylène glycol – et ß galactosidase +, ce ne sont pas des Salmonella : absence de Salmonella dans les selles
Pas de culture sur Campylosel = absence de Campylobacter.
Les petites colonies lactose – sur BCP et les petites colonies glucides + sur Hektoen sont probablement les Yersinia (Yersinia enterocolitica est lactose – et saccharose +.
| FIN DE L’ÉTUDE DE CAS |