Coproculture
Un homme de 35 ans souffre de diarrhées accompagnées de douleurs abdominales et de vomissements. Il a de la fièvre. Il n’a pas voyagé récemment.
Le médecin prescrit une coproculture standard.
| Aspect des selles |
Muco-purulentes |
| Gram objectif X100 |
Flore polymicrobienne constituée de 70% de Gram – et 30% de Gram + |
| EF objectif x40 |
Leucocytes : environ 15 /champ
Hématies : 5/champ Pas de bacille mobile par ciliature polaire, pas de levures |
Questions
| Pour faire apparaitre la réponse, cliquez sur la question ! |
La flore est équilibrée.
La présence de nombreux leucocytes oriente vers une diarrhée inflammatoire.
Il ne semble pas qu’il y ait des Campylobacter ou des Vibrio car il n’a pas été observé de bactérie présentant une mobilité « en vol de moucheron ». Mais cela reste à confirmer en poursuivant la recherche des Campylobacter par la culture. En revanche, il n’était pas nécessaire d’ensemencer des milieux sélectifs des Vibrio car il n’y a pas de notion de voyage récent.
Pas de mycose.
| Liens utiles |
| Coproculture |
| BCP à 37°C | Très nombreuses colonies jaunes et quelques colonies incolores |
| Hektoen à 37°C | Très nombreuses colonies saumon et quelques colonies vertes sans centre noir, mal isolées |
| Yersinia CIN à 30°C | Pas de culture |
| CAMPYLOSEL à 37°C sous atmosphère microaérobie | Pas de culture |
| Bouillon Rappaport à 37°C | Présence d’un trouble |
Les colonies vertes sans centre noir sur Hektoen sont lactose -, saccharose -, salicine – et H2S -. Elles peuvent être des Salmonella ou des Shigella. En revanche, les colonies saumon utilisent un des glucides et ainsi ne peuvent être des Salmonella ou des Shigella.
Réaliser au moins 5 tests uréase rapide comprenant chacun une colonie suspecte (colonies vertes à centre noir sur Hektoen). On peut également identifier par spectrométrie de masse MALDI TOF plusieurs de ces colonies suspectes (au moins 5).
Remise à l’étuve des milieux CIN et Campylosel.
Faire un isolement sur milieu chromogène sélectif des Salmonella de la culture en bouillon Rappaport. Dans le cas présent c’est la gélose SM2 qui a été ensemencée.
| Les 5 colonies testées sont uréase -. |
Ajouter 5 mL d’eau distillée stérile à une suspension « uréase négative » et, ensemencer avec, une galerie d’identification associée à un contrôle de pureté sur BCP. Dans le cas présent, le technicien a choisi une API 20E.
Il faut également ensemencer une gélose nutritive en pente pour un éventuel sérotypage.
|
Pas de culture sur Yersinia CIN et Campylosel Culture sur le milieu SM2 avec présence de nombreuses colonies bleues et de rares colonies incolores sur le milieu SM2. Identification de la colonie suspecte sur Hektoen Lecture du contrôle de pureté = nombreuses petites colonies incolores et quelques colonies jaunes plus grosses Le technicien décide de lire cette galerie. |
Pas de culture sur Yersinia CIN après 48h : Absence de Yersinia dans les selles.
Pas de culture sur Campylosel après 48h : Absence de Campylobacter dans les selles.
Absence de colonies mauves sur SM2 : Absence de Salmonella dans les selles
| Document utile |
|
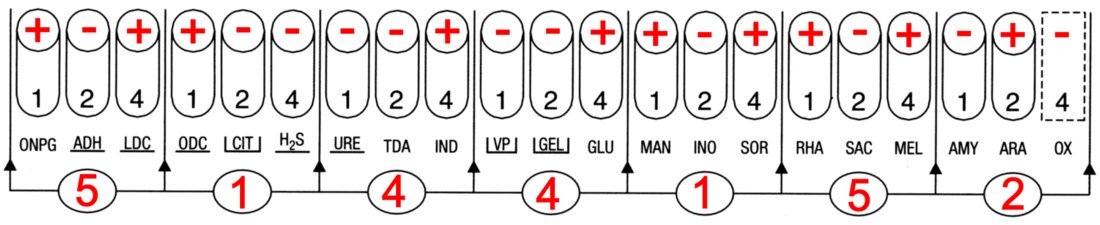
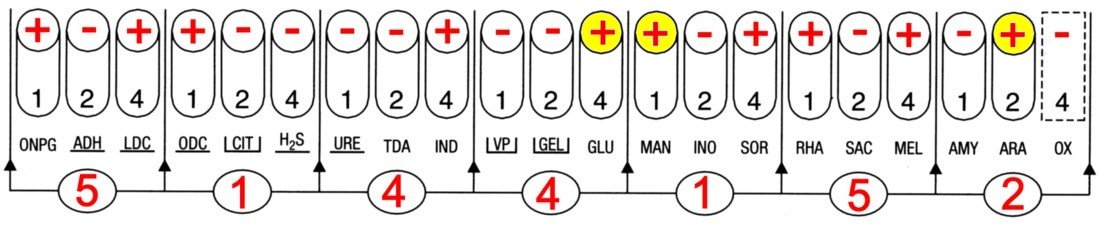
La galerie API 20E présente un profil caractéristique de Escherichia coli.
| Liens utiles |
| Identifieur « api » d’UPBM le Lab (un grand merci à Antoine Gaudin et Jean-Noël Joffin !) |
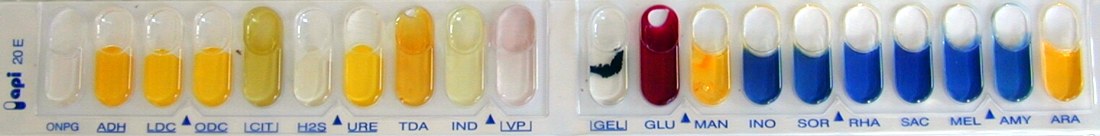
La colonie suspecte sur Hektoen a été identifiée comme un Escherichia coli. Mais cette identification n’est pas valable car le contrôle de pureté présente deux types de colonies. La galerie a donc été ensemencée avec une suspension contenant deux germes.
Descendez bien plus bas pour voir la suite du questionnaire.
| ATTENTION ! Ce n’est pas fini, la suite est plus bas. » |
Il doit ensemencer, avec une suspension préparée à partir des colonies incolores sur le contrôle de pureté, une nouvelle galerie API20E + contrôle de pureté sur BCP + gélose inclinée pour un éventuel sérotypage.
|
Lecture du contrôle de pureté sur BCP = un seul type de colonie (petites colonies incolores) |
| Document utile |
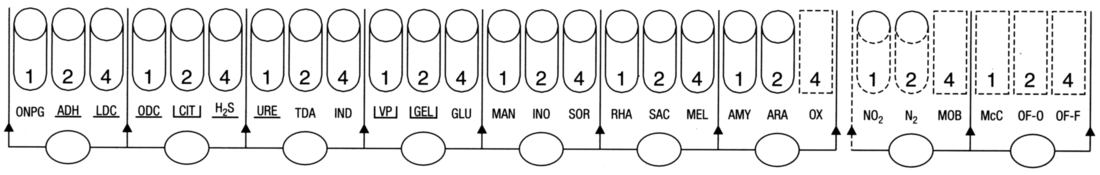 Fiche technique de la galerie API 20E |
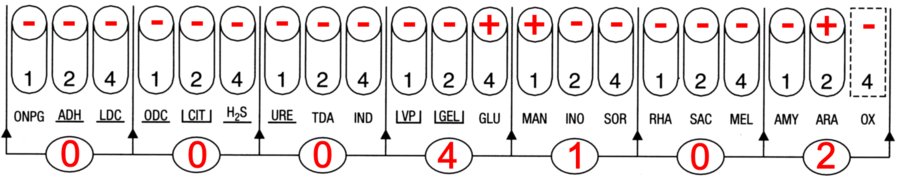
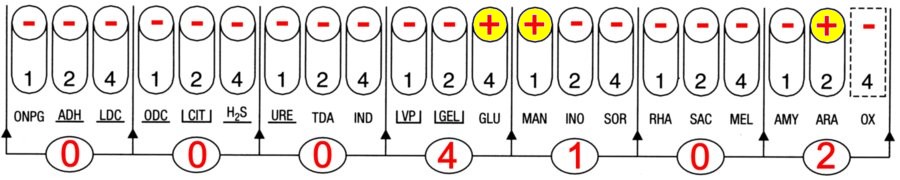
La galerie API 20E identifie Shigella spp avec une probabilité de 72,1%. Ensuite vient Escherichia coli avec une probabilité de 17,9 % puis Yersinia pestis avec une probabilité de 9,5%.
| Liens utiles |
| Identifieur « api » d’UPBM le Lab (un grand merci à Antoine Gaudin et Jean-Noël Joffin !) |
S’il s’agit bien d’une Shigella (probabilité 72,1%), le sujet présente alors une infection intestinale à Shigella spp.
Shigella spp présente très peu de caractères positifs en API 20E et le peu qu’elle possède sont en commun avec Escherichia coli. Ainsi la présence de Shigella dans la suspension ne perturbe pas le profil caractéristique d’un Escherichia coli.
Le technicien aurait pu conclure à une absence de Shigella dans les selles. En prélevant les colonies de Shigella pour réaliser le test uréase rapide, il a malencontreusement récupéré un petit peu d’Escherichia coli mais suffisamment pour perturber l’identification.
Ces résultats montrent l’importance d’une observation attentive du contrôle de pureté. Si le milieu utilisé n’avait pas été lactosé, la distinction entre les colonies de Shigella (lactose -) et celles d’E. coli (lactose +) aurait été plus difficile avec un risque de diagnostic erroné. Le milieu BCP convient pour le contrôle de pureté car il est à la fois non sélectif et lactosé.
L’identification n’est pas certaine et dans le cas présent, il est nécessaire de s’assurer que c’est une Shigella en pratiquant un sérotypage.
|
Pas d’agglutination en eau physiologique, Sérum D : pas d’agglutination Sérum polyvalent B : agglutination |
Pas d’agglutination en eau physiologique : la souche n’est pas autoagglutinable
Pas d’agglutination avec le sérum D : la souche n’appartient pas à l’espèce Shigella sonnei
Agglutination avec le sérum B : la souche est une Shigella flexneri.
| FIN DE L’ÉTUDE DE CAS |