Suite aux recommandations de la HAS, les méthodes qui utilisent la culture cellulaire, la détection d’antigènes par immunofluorescence ou ELISA et la détection du génome de Chlamydia trachomatis par biologie moléculaire sans amplification ont été supprimées de la Nomenclature Des Actes De Biologie Médicale (NABM) le 4 novembre 2011. En effet, ces méthodes manquent de sensibilité ou de spécificité.
Désormais seule une technique est inscrite à la NABM. Il s’agit de la recherche d’ADN ou d’ARN de Chlamydia trachomatis par amplification génique in vitro.
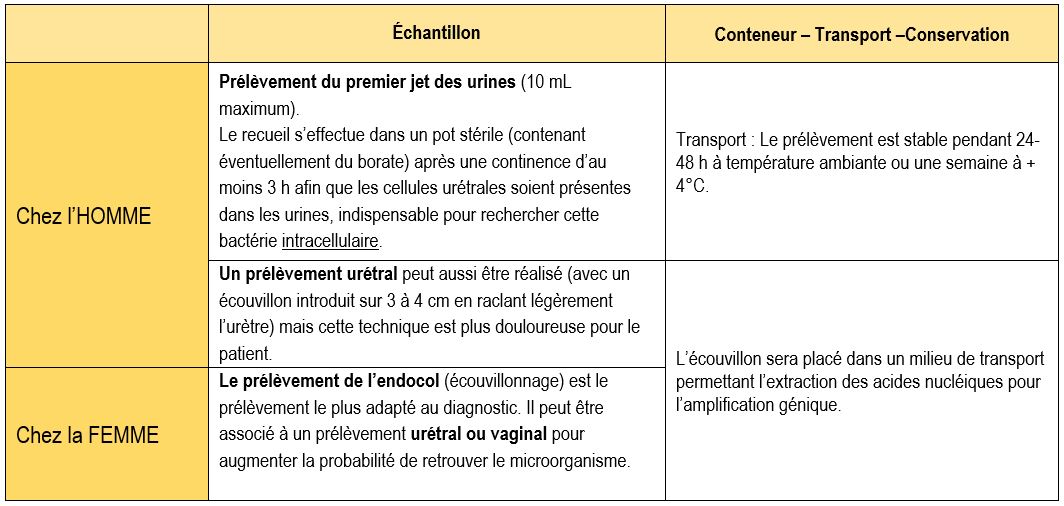
Prélèvement et transport
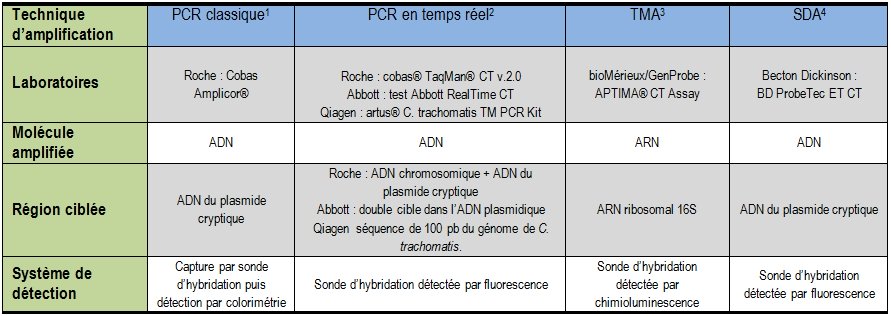
Tests d’amplification génique
Les trousses actuelles présentent à la fois une bonne sensibilité (> 95%, c’est-à-dire moins de 5% de faux négatifs) et une bonne spécificité (> 95%, c’est-à-dire moins de 5% de faux positifs).
Les cibles d’amplification peuvent être :
- une séquence d’ADN localisée dans un plasmide appelé « plasmide cryptique » qui est commun à tous les sérovars de Chlamydia trachomatis (plasmide présent en 7 à 10 exemplaires par bactérie)
- des fragments d’ARNr.
Des contrôles internes d’amplification permettent de s’assurer que l’échantillon ne contient pas d’inhibiteurs de l’amplification.
On a identifié, en Suède en 2006, un variant génétique de Chlamydia trachomatis qui présente une délétion de 350 pb sur le plasmide cryptique. Or les amorces du test de détection par PCR se lient à la région du plasmide qui a disparu. De ce fait, ce variant n’est plus détectable par les tests ciblant seulement cette région.
Tableau 3 : les différentes techniques d’amplification génique appliquées à Chlamydia trachomatis
Les méthodes d’amplification génique sont présentées sur d’autres pages de ce site :
- PCR (Polymerase Chain Reaction)
- PCR en temps réel
- TMA (Transcription Mediated Amplification)
- SDA (Stand Displacement Amplification)