La PCR (Polymerase Chain Reaction) permet d’amplifier in vitro une région spécifique d’un acide nucléique donné.
Elle se déroule par cycles composés de trois étapes (Cf. fig. 1):
- étape 1 : dénaturation (à 95°c) des doubles brins d’ADN pour donner des simples brins
- étape 2 : hybridation (environ 55°C) d’un couple d’oligonucléotides amorces (amorce A et amorce B) qui délimitent la région d’ADN à amplifier
- étape 3 : l’élongation par une ADN polymérase thermorésistante (environ 72°C) à partir des extrémités 3’OH des amorces et ainsi la formation de brins complémentaires.
Les produits obtenus à la fin de chaque cycle servent de matrice pour le cycle suivant, l’amplification est donc exponentielle.
Les tubes contenant le mélange réactionnel sont placés dans un appareil permettant de faire varier la température au cours du temps et appelé thermocycleur.
Le mélange réactionnel contient :
-
- tampon
- magnésium
- des désoxyribonucléotides : dATP, dCTP, dGTP et dTTP
- une amorce A et une amorce B
- une ADN polymérase thermorésistante
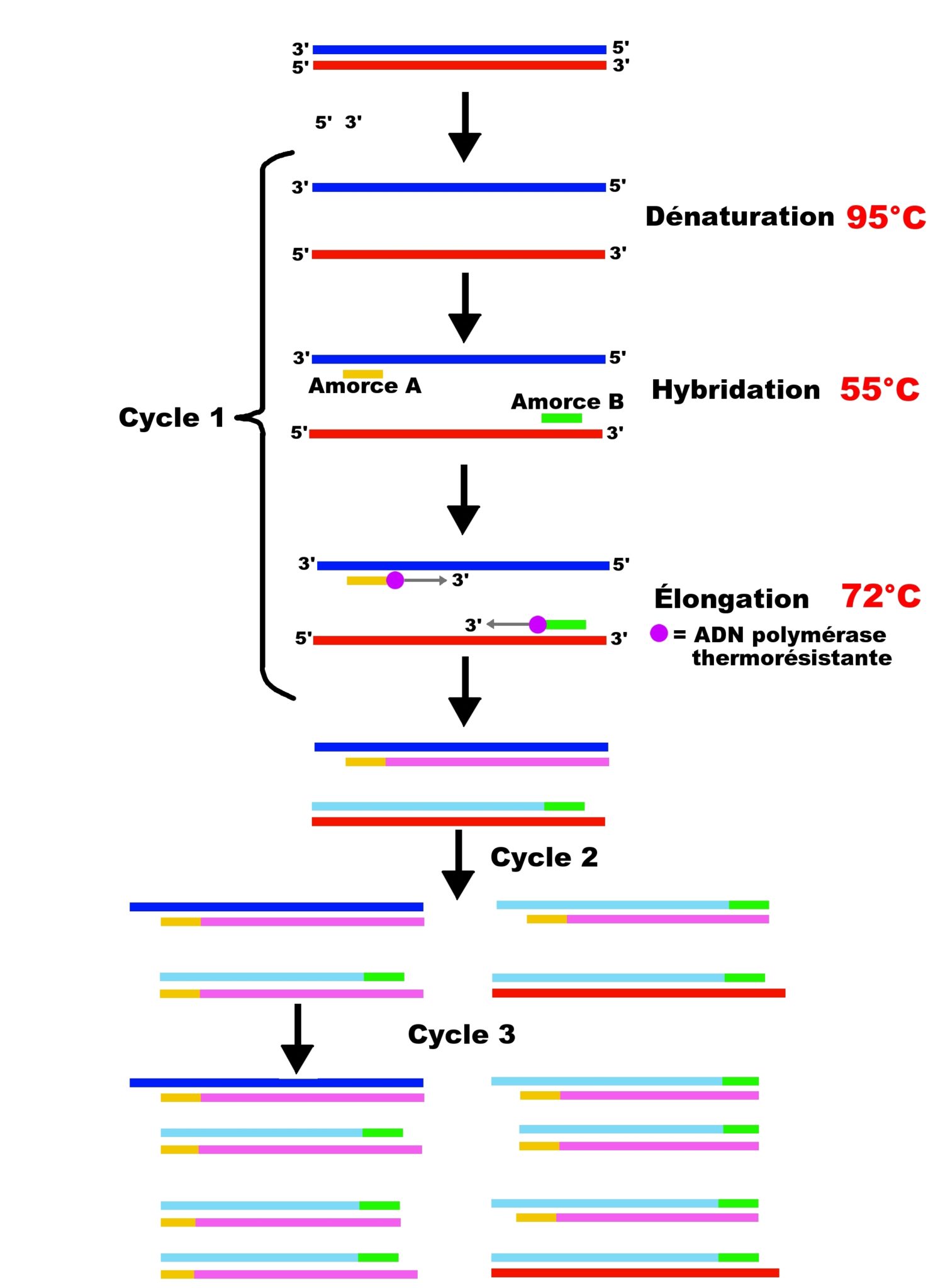
Fig. 1 : Chronologie de la PCR
Cobas Amplicor®
Le gène cible se trouve sur le « plasmide cryptique » (il y a environ de 7 à 10 copies de ce plasmide/bactérie).
Après une trentaine de cycle d’amplification on obtient plusieurs millions de copies de la séquence cible (amplicons).
Ces amplicons sont capturés par des sondes spécifiques revêtant une microplaque. La présence de biotine sur les amorces utilisées lors de l’amplification permet alors de révéler les amplicons par un conjugué avidine-peroxydase en présence d’un substrat chromogène. Cf. fig. 2.
Fig. 2 : Révélation des amplicons (trousse Cobas Amplicor)
