Le test APTIMA® CT Assay de bioMérieux/GenProbe
La TMA est une méthode d’amplification isotherme d’ARN. La séquence amplifiée appartient à l’ARN16S.
Cette méthode est très sensible car l’ARN 16S est présent à environ 1000 copies/bactérie
Le mélange réactionnel contient :
- du tampon
- du magnésium
- des ribonucléotides : ATP, CTP, GTP et UTP
- des désoxyribonucléotides : dATP, dCTP, dGTP et dTTP
- une amorce A comportant à son extrémité 5’ une séquence non complémentaire de la cible et qui correspond à un site d’initiation de la transcription de l’ARN polymérase du phage T7.
- une seconde amorce B
- l’ARN polymérase du phage T7
- une transcriptase inverse particulière, appelée Moloney murine leukemia virus RT (M-MLV-RT). Comme toutes les transcriptases inverses, c’est une ADN polymérase capable de synthétiser un brin d’ADN complémentaire (ADNc) en prenant un ARN comme matrice. La particularité de cette transcriptase inverse est de posséder également une activité RNase.
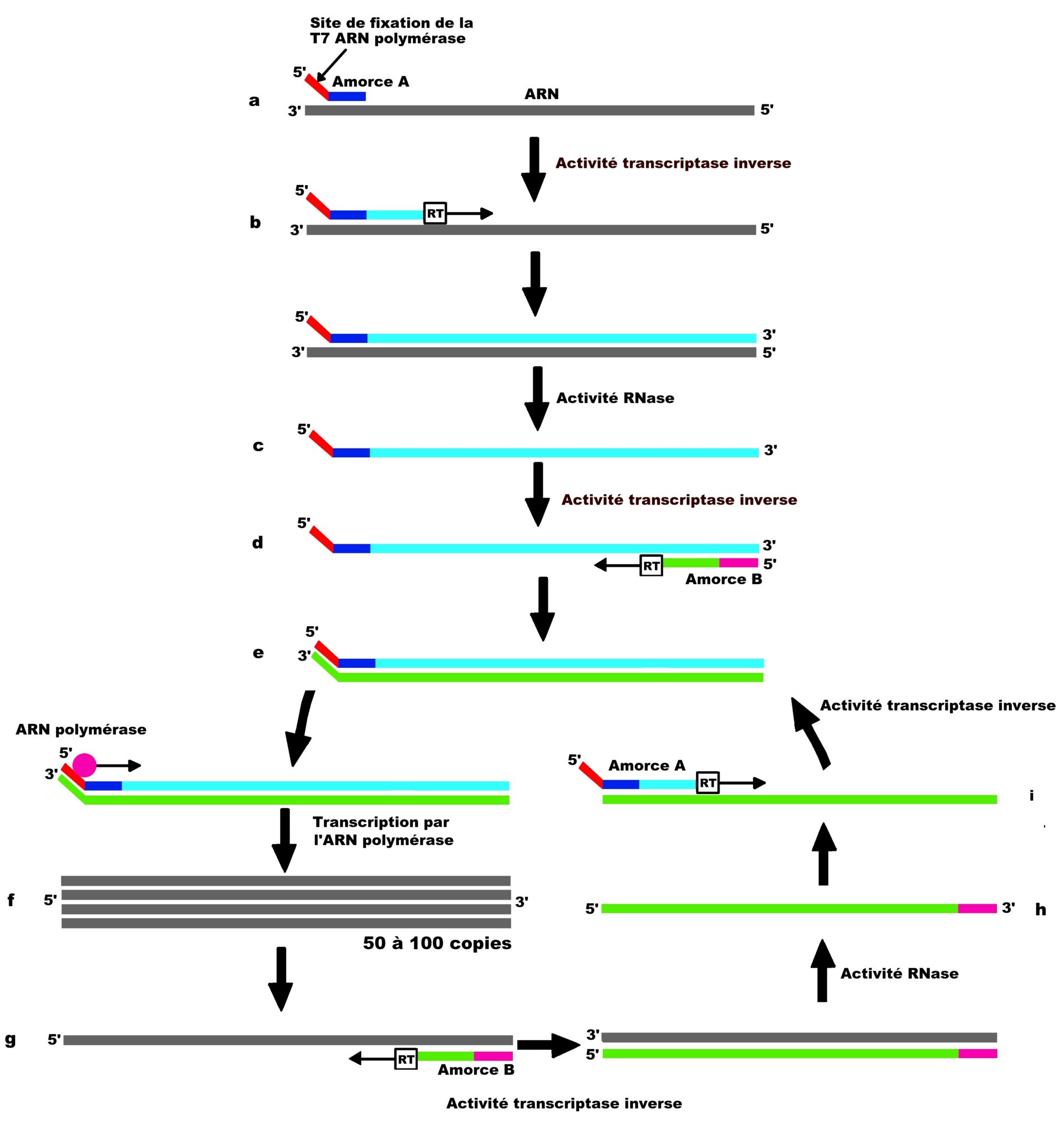
Cette méthode s’effectue en deux temps : la synthèse d’un ADN double brin transcriptible en ARN puis une phase d’amplification (Fig.5)
Premier temps : la synthèse d’un ADN double brin transcriptible en ARN
L’amorce A qui contient à son extrémité 5’ un site de fixation de l’ARN polymérase du phage T7 s’hybride à l’ARN cible (étape a). La transcriptase inverse (RT) réalise l’élongation de l’amorce A par incorporation de dNTP (étape b) permettant d’obtenir un hybride ARN/ADN. L’activité RNase de RT dégrade ensuite l’ARN de cet hybride et libère un ADN simple brin (étape c). L’amorce B peut alors s’hybrider à ce monobrin d’ADN (étape d). Son élongation par la transcriptase inverse (RT) permet d’obtenir un ADN double brin comportant grâce à l’amorce A, un site promoteur de l’ARN polymérase du page T7 (étape e).
Deuxième temps : la phase d’amplification
L’ARN polymérase se fixe sur l’ADN double brin et transcrit de très nombreux ARN (étape f). Les ARN synthétisés servent à leur tour de matrice pour la synthèse d’un ADN double brin avec hybridation de l’amorce B et son élongation par la transcriptase inverse (étape g), dégradation du brin d’ARN (étape h), hybridation de l’amorce A et son élongation par la transcriptase inverse (étape i) pour obtenir de nouveau un double brin d’ADN qui subira un nouveau cycle.
Fig.5
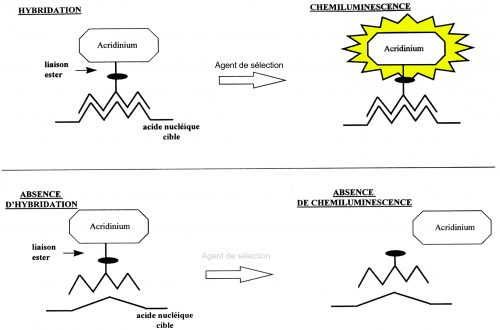
La détection des amplicons utilise la méthode HPA (Hybridization Protect Assay)
Une sonde marquée à l’aide d’une molécule d’ester d’acridinium reconnait une séquence spécifique de l’amplicon (ARN).
Après une phase permettant l’hybridation, on ajoute de la soude au mélange réactionnel. La solution de sélection (solution tamponnée de borate) rompt seulement les liaisons ester entre l’acridinium et les sondes non hybridées. L’acridinium libre n’est plus chimioluminescent. Ainsi, seules les sondes hybridées avec la région cible donnent un signal chimioluminescent. (Cf. fig. 6)
Fig.6